

Желейный рулет или швейцарский рулет — белковая укладка или сверхвторичная структура, состоящая из восьми бета-нитей, расположенных в двух четырёхнитевых листах. Название структуры, введённое Джейн С. Ричардсон в 1981 году, отражает его сходство с желейным или швейцарским пирогом[2]. Укладка является продолжением мотива «Греческий ключ» и иногда считается формой бета-цилиндра. Она очень часто встречается в вирусных белках, особенно в вирусных капсидных белках[3][4]. Взятые вместе, желейный рулет и греческий ключ составляют около 30 % всех бета-белков, аннотированных в базе данных структурной классификации белков (SCOP)[5].
Структура
Основная структура желейного рулета состоит из восьми бета-нитей, расположенных в двух четырёхцепочечных антипараллельных бета-листах, которые упаковываются вместе через гидрофобную поверхность раздела. Цепи традиционно обозначаются буквами от B до I по той исторической причине, что первая решённая структура, капсидный белок желе из вируса кустистости томатов, имела дополнительную цепь A за пределами общего ядра укладки[6][7]. Листы состоят из прядей BIDG и CHEF, сложенных таким образом, что прядь B упаковывает прядь напротив C, I напротив H и т. д.[4][8]
Вирусные белки
Большое количество вирусов строят свои капсиды из белков, содержащих либо одинарную, либо двойную укладку желейного рулета. Считается, что эта общая капсидная архитектура отражает древние эволюционные отношения, возможно, восходящие к последнему универсальному общему предку (LUCA) клеточной жизни[8][9][10]. Другие вирусные линии используют эволюционно неродственные белки для создания закрытых капсидов, которые, вероятно, независимо эволюционировали, по крайней мере, дважды[9][11] и, возможно, много раз, со связями с белками клеточного происхождения[12].
Протеины капсида с одиночным желейным рулетом
Белки с одним желейным рулетом в капсиде (JRC) обнаружены по крайней мере в шестнадцати различных вирусных семействах, в основном с икосаэдрической капсидной структурой и включающими как РНК-вирусы, так и ДНК-вирусы[13]. Многие вирусы с одноцепочечным капсидом представляют собой одноцепочечные РНК- положительные вирусы. Две группы двухцепочечных ДНК-вирусов с одноцепочечными капсидами JRC — это Papillomaviridae и Polyomaviridae, обе из которых имеют довольно маленькие капсиды; у этих вирусов архитектура собранного капсида ориентирует ось желейного рулета параллельно или «горизонтально» относительно поверхности капсида[11]. Масштабный анализ компонентов вирусного капсида показал, что одиночный горизонтальный желейный рулет является наиболее распространенной укладкой среди белков капсида, на долю которого приходится около 28 % известных примеров[12].
Другая группа вирусов использует в капсидах единичные белки желейного рулета, но в вертикальной, а не горизонтальной ориентации. Эти вирусы эволюционно родственны большой группе вирусов с двойным желейным рулетом, известной как PRD1 — вирусной линии аденовируса, со сходной архитектурой капсида, реализованной путем сборки двух отдельных основных капсидных белков с укладкой желейного рулета, экспрессируемых из разных генов[14][15]. Эти одиночные вирусы с вертикальным желейным рулетом составляют таксон Helvetiavirae[16]. Известные вирусы с вертикальными капсидами в виде одинарных желейных рулетов заражают экстремофильных прокариот[14][12].
Двойной желейный рулет

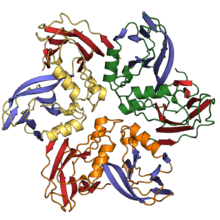
Капсидные белки двойного желейного рулета состоят из двух одиночных укладок желейного рулета, соединенных короткой линкерной областью. Они обнаруживаются как в двухцепочечных ДНК-вирусах, так и в одноцепочечных ДНК-вирусах по крайней мере десяти различных вирусных семейств, включая вирусы, которые инфицируют все домены жизни и охватывают большой диапазон размеров капсидов[4][11][18]. В архитектуре капсида с двойным желейным рулетом его ось ориентирована перпендикулярно или «вертикально» относительно поверхности капсида[19].
Считается, что белки с двойным желейным рулетом произошли от белков с одиночным желейным рулетом путем дупликации генов[11][19]. Вполне вероятно, что вирусы с вертикальным одинарным желейным рулетом представляют собой переходную форму, и что белки капсида вертикального и горизонтального желейного рулета имеют независимое эволюционное происхождение от предковых клеточных белков[12]. Степень структурного сходства капсидов вирусов двойного желейного рулета привела к выводу, что эти вирусы, вероятно, имеют общее эволюционное происхождение, несмотря на их разнообразие по размеру и диапазону хозяев; это стало известно как линия PRD1 — аденовируса (Bamfordvirae)[19][16][20][21]. Многие члены этой группы были идентифицированы с помощью метагеномики, и в некоторых случаях они практически не имеют общих вирусных генов[12][22]. Хотя большинство членов этой группы имеют икосаэдрическую геометрию капсида, несколько семей, таких как Poxviridae и Ascoviridae имеют овальные или кирпичные формы зрелых вирионов; Поксвирусы, такие как Vaccinia, претерпевают резкие конформационные изменения, опосредованные белками двойного желейного рулета во время созревания и, вероятно, происходят от икосаэдрического предка[11][23].
Белки двойного желейного рулета в клеточных белках не наблюдались; они кажутся уникальными для вирусов[11]. По этой причине обнаружение явной гомологии с белками двойного желейного рулета в последовательностях мобильных элементов - полинтонов, широко распространенных в геномах эукариот, считается доказательством тесной эволюционной связи этих генетических элементов с вирусами[24].
Некапсидные вирусные белки
Одиночные желейные рулеты также встречаются в некапсидных вирусных белках, включая второстепенные компоненты собранного вириона, а также в невирионных белках, таких как полиэдрин[11].
Клеточные белки
В то время как двойные желейные рулеты не встречаются в белках клеточного происхождения, встречаются одинарные желейные рулеты[11][12]. Одним из таких классов клеточных белков являются нуклеоплазмины, которые служат в качестве молекулярных белков-шаперонов для сборки гистонов в нуклеосомы. N-концевой домен нуклеоплазмина имеет единственную укладку желейного рулета и собран в пентамер[25]. С тех пор о подобных структурах сообщалось в дополнительных группах белков ремоделирования хроматина[26]. Мотивы желейного рулета с идентичной связностью с бета-слоями также обнаруживаются в лигандах фактора некроза опухолей[27] и белках бактерии Yersinia pseudotuberculosis, которые принадлежат к классу вирусных и бактериальных белков, известных как суперантигены[28][29].
В более широком смысле члены чрезвычайно разнообразного суперсемейства купинов также часто описываются как желейные рулеты; хотя общее ядро доменной структуры купина содержит только шесть бета-цепей, многие купины имеют восемь[30]. Примеры включают негем- диоксигеназные ферменты[31][32] и гистоновые деметилазы семейства JmjC[33][34].
Эволюция
Сравнительные исследования белков, классифицируемых как желейный рулет и «Греческий ключ», предполагают, что ключевые протеины Греческого ключа возникли значительно раньше, чем их более топологически сложные аналоги в желейном рулете[5]. Структурные биоинформатические исследования, сравнивающие белки вирусного капсидного желейного рулета с другими белками известной структуры, показывают, что белки капсида образуют хорошо разделенный кластер, что позволяет предположить, что они подвержены определённому набору эволюционных ограничений[4]. Одной из наиболее примечательных особенностей белков в виде желейного рулета вирусного капсида является их способность образовывать олигомеры в виде повторяющейся мозаичной структуры с образованием замкнутой белковой оболочки; клеточные белки, наиболее похожие по укладке и топологии, в большинстве своем также являются олигомерами[4]. Было высказано предположение, что вирусные капсидные белки желейного рулета произошли из клеточных желейных рулетов, возможно, в нескольких независимых случаях, на самых ранних стадиях клеточной эволюции[12].
История и номенклатура
Название «желейный рулет» было впервые использовано для структуры, созданной Джейн С. Ричардсон в 1981 году на основе греческого ключевого мотива, и было призвано отразить сходство структуры с желейным или швейцарским пирогом[2]. Структура получила множество описательных названий, в том числе: клин, бета-ствол и бета-ролл. Края двух листов не пересекаются, чтобы сформировать регулярные узоры водородных связей, и поэтому его часто не считают истинным бета-цилиндром[3] хотя этот термин широко используется при описании архитектуры вирусного капсида[14][15]. Клеточные белки, содержащие структуры, похожие на желейный рулет, можно описать как купиновую складку, складку JmjC или двухцепочечную бета-спираль[32].
См. также
Примечания
- ↑ 1 2 Steven B. Larson, John S. Day, Alexander McPherson. Satellite tobacco mosaic virus refined to 1.4 Å resolution // Acta Crystallographica Section D Biological Crystallography. — 2014-09-01. — Т. 70, вып. 9. — С. 2316–2330. — ISSN 1399-0047. — doi:10.1107/S1399004714013789. Архивировано 20 июня 2022 года.
- ↑ 1 2 Advances in protein chemistry. Volume 34. — New York: Academic Press, 1981. — 1 online resource (ixi, 371 pages) с. — ISBN 978-0-08-058207-8, 0-08-058207-9, 0-12-034234-0, 978-0-12-034234-1.
- ↑ 1 2 Gareth Chelvanayagam, Jaap Heringa, Patrick Argos. Anatomy and evolution of proteins displaying the viral capsid jellyroll topology (англ.) // Journal of Molecular Biology. — 1992-11. — Vol. 228, iss. 1. — P. 220–242. — doi:10.1016/0022-2836(92)90502-B. Архивировано 15 июня 2022 года.
- ↑ 1 2 3 4 5 Shanshan Cheng, Charles L. Brooks. Viral Capsid Proteins Are Segregated in Structural Fold Space (англ.) // PLoS Computational Biology / Dennis R. Livesay. — 2013-02-07. — Vol. 9, iss. 2. — P. e1002905. — ISSN 1553-7358. — doi:10.1371/journal.pcbi.1002905.
- ↑ 1 2 Hannah Edwards, Sanne Abeln, Charlotte M. Deane. Exploring Fold Space Preferences of New-born and Ancient Protein Superfamilies (англ.) // PLoS Computational Biology / Christine A. Orengo. — 2013-11-14. — Vol. 9, iss. 11. — P. e1003325. — ISSN 1553-7358. — doi:10.1371/journal.pcbi.1003325.
- ↑ S. C. Harrison, A. J. Olson, C. E. Schutt, F. K. Winkler, G. Bricogne. Tomato bushy stunt virus at 2.9 Å resolution (англ.) // Nature. — 1978-11. — Vol. 276, iss. 5686. — P. 368–373. — ISSN 1476-4687 0028-0836, 1476-4687. — doi:10.1038/276368a0. Архивировано 17 октября 2022 года.
- ↑ Michael G. Rossmann, Celerino Abad-Zapatero, Mathur R.N. Murthy, Lars Liljas, T. Alwyn Jones. Structural comparisons of some small spherical plant viruses (англ.) // Journal of Molecular Biology. — 1983-04. — Vol. 165, iss. 4. — P. 711–736. — doi:10.1016/S0022-2836(83)80276-9. Архивировано 5 августа 2020 года.
- ↑ 1 2 Michael G. Rossmann, Celerino Abad-Zapatero, Mathur R. N. Murthy, Lars Liljas, T. Alwyn Jones. Structural comparisons of some small spherical plant viruses (англ.) // Journal of Molecular Biology. — 1983-04-25. — Vol. 165, iss. 4. — P. 711–736. — ISSN 0022-2836. — doi:10.1016/S0022-2836(83)80276-9.
- ↑ 1 2 Patrick Forterre, David Prangishvili. The origin of viruses (англ.) // Research in Microbiology. — 2009-09. — Vol. 160, iss. 7. — P. 466–472. — doi:10.1016/j.resmic.2009.07.008. Архивировано 26 октября 2022 года.
- ↑ Edward C. Holmes. What Does Virus Evolution Tell Us about Virus Origins? (англ.) // Journal of Virology. — 2011-06. — Vol. 85, iss. 11. — P. 5247–5251. — ISSN 1098-5514 0022-538X, 1098-5514. — doi:10.1128/JVI.02203-10. Архивировано 14 марта 2022 года.
- ↑ 1 2 3 4 5 6 7 8 Mart Krupovic, Dennis H Bamford. Double-stranded DNA viruses: 20 families and only five different architectural principles for virion assembly (англ.) // Current Opinion in Virology. — 2011-08. — Vol. 1, iss. 2. — P. 118–124. — doi:10.1016/j.coviro.2011.06.001. Архивировано 16 октября 2022 года.
- ↑ 1 2 3 4 5 6 7 Mart Krupovic, Eugene V. Koonin. Multiple origins of viral capsid proteins from cellular ancestors (англ.) // Proceedings of the National Academy of Sciences. — 2017-03-21. — Vol. 114, iss. 12. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.1621061114. Архивировано 3 сентября 2022 года.
- ↑ Mart Krupovic. Networks of evolutionary interactions underlying the polyphyletic origin of ssDNA viruses (англ.) // Current Opinion in Virology. — 2013-10. — Vol. 3, iss. 5. — P. 578–586. — doi:10.1016/j.coviro.2013.06.010. Архивировано 16 октября 2022 года.
- ↑ 1 2 3 David Gil-Carton, Salla T. Jaakkola, Diego Charro, Bibiana Peralta, Daniel Castaño-Díez. Insight into the Assembly of Viruses with Vertical Single β-barrel Major Capsid Proteins (англ.) // Structure. — 2015-10. — Vol. 23, iss. 10. — P. 1866–1877. — doi:10.1016/j.str.2015.07.015. Архивировано 11 июля 2022 года.
- ↑ 1 2 Isaac Santos-Pérez, Diego Charro, David Gil-Carton, Mikel Azkargorta, Felix Elortza. Structural basis for assembly of vertical single β-barrel viruses (англ.) // Nature Communications. — 2019-12. — Vol. 10, iss. 1. — P. 1184. — ISSN 2041-1723. — doi:10.1038/s41467-019-08927-2. Архивировано 3 сентября 2022 года.
- ↑ 1 2 Eugene V Koonin, Valerian V. Dolja, Mart Krupovic, Arvind Varsani, Yuri I Wolf. Create a megataxonomic framework, filling all principal taxonomic ranks, for DNA viruses encoding vertical jelly roll-type major capsid proteins. — 2019. — doi:10.13140/RG.2.2.14886.47684.
- ↑ 1 2 Nicola G.A. Abrescia, Jonathan M. Grimes, Hanna M. Kivelä, Rene Assenberg, Geoff C. Sutton. Insights into Virus Evolution and Membrane Biogenesis from the Structure of the Marine Lipid-Containing Bacteriophage PM2 (англ.) // Molecular Cell. — 2008-09. — Vol. 31, iss. 5. — P. 749–761. — doi:10.1016/j.molcel.2008.06.026. Архивировано 21 октября 2022 года.
- ↑ Elina Laanto, Sari Mäntynen, Luigi De Colibus, Jenni Marjakangas, Ashley Gillum. Virus found in a boreal lake links ssDNA and dsDNA viruses (англ.) // Proceedings of the National Academy of Sciences. — 2017-08. — Vol. 114, iss. 31. — P. 8378–8383. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.1703834114. Архивировано 3 сентября 2022 года.
- ↑ 1 2 3 Mart Krupovič, Dennis H. Bamford. Virus evolution: how far does the double β-barrel viral lineage extend? (англ.) // Nature Reviews Microbiology. — 2008-12. — Vol. 6, iss. 12. — P. 941–948. — ISSN 1740-1534 1740-1526, 1740-1534. — doi:10.1038/nrmicro2033. Архивировано 3 сентября 2022 года.
- ↑ Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic, Arvind Varsani, Yuri I. Wolf. Global Organization and Proposed Megataxonomy of the Virus World (англ.) // Microbiology and Molecular Biology Reviews. — 2020-05-20. — Vol. 84, iss. 2. — P. e00061–19. — ISSN 1098-5557 1092-2172, 1098-5557. — doi:10.1128/MMBR.00061-19. Архивировано 26 июля 2021 года.
- ↑ Peter J. Walker, Stuart G. Siddell, Elliot J. Lefkowitz, Arcady R. Mushegian, Evelien M. Adriaenssens. Changes to virus taxonomy and the Statutes ratified by the International Committee on Taxonomy of Viruses (2020) (англ.) // Archives of Virology. — 2020-11. — Vol. 165, iss. 11. — P. 2737–2748. — ISSN 1432-8798 0304-8608, 1432-8798. — doi:10.1007/s00705-020-04752-x.
- ↑ “Vast diversity of prokaryotic virus genomes encoding double jelly-roll major capsid proteins uncovered by genomic and metagenomic sequence analysis”. Virology Journal. 15 (1): 67. April 2018. DOI:10.1186/s12985-018-0974-y. PMID 29636073.
- ↑ Mohammad W. Bahar, Stephen C. Graham, David I. Stuart, Jonathan M. Grimes. Insights into the Evolution of a Complex Virus from the Crystal Structure of Vaccinia Virus D13 (англ.) // Structure. — 2011-07. — Vol. 19, iss. 7. — P. 1011–1020. — doi:10.1016/j.str.2011.03.023. Архивировано 18 августа 2022 года.
- ↑ Mart Krupovic, Dennis H Bamford, Eugene V Koonin. Conservation of major and minor jelly-roll capsid proteins in Polinton (Maverick) transposons suggests that they are bona fide viruses (англ.) // Biology Direct. — 2014. — Vol. 9, iss. 1. — P. 6. — ISSN 1745-6150. — doi:10.1186/1745-6150-9-6. Архивировано 15 июня 2022 года.
- ↑ Shuchismita Dutta, Ildikó V. Akey, Colin Dingwall, Kari L. Hartman, Tom Laue. The Crystal Structure of Nucleoplasmin-Core (англ.) // Molecular Cell. — 2001-10. — Vol. 8, iss. 4. — P. 841–853. — doi:10.1016/S1097-2765(01)00354-9. Архивировано 17 июня 2022 года.
- ↑ Christian Edlich-Muth, Jean-Baptiste Artero, Phil Callow, Marcin R. Przewloka, Aleksandra A. Watson. The Pentameric Nucleoplasmin Fold Is Present in Drosophila FKBP39 and a Large Number of Chromatin-Related Proteins (англ.) // Journal of Molecular Biology. — 2015-05. — Vol. 427, iss. 10. — P. 1949–1963. — doi:10.1016/j.jmb.2015.03.010. Архивировано 19 июня 2022 года.
- ↑ Jean-Luc Bodmer, Pascal Schneider, Jürg Tschopp. The molecular architecture of the TNF superfamily (англ.) // Trends in Biochemical Sciences. — 2002-01. — Vol. 27, iss. 1. — P. 19–26. — doi:10.1016/S0968-0004(01)01995-8. Архивировано 15 июня 2022 года.
- ↑ Roberta Donadini, Chu Wai Liew, Ann H.Y. Kwan, Joel P. Mackay, Barry A. Fields. Crystal and Solution Structures of a Superantigen from Yersinia pseudotuberculosis Reveal a Jelly-Roll Fold (англ.) // Structure. — 2004-03. — Vol. 12, iss. 1. — P. 145–156. — doi:10.1016/j.str.2003.12.002.
- ↑ John D. Fraser, Thomas Proft. The bacterial superantigen and superantigen-like proteins (англ.) // Immunological Reviews. — 2008-10. — Vol. 225, iss. 1. — P. 226–243. — doi:10.1111/j.1600-065X.2008.00681.x. Архивировано 3 сентября 2022 года.
- ↑ Sawsan Khuri, Freek T. Bakker, Jim M. Dunwell. Phylogeny, Function, and Evolution of the Cupins, a Structurally Conserved, Functionally Diverse Superfamily of Proteins (англ.) // Molecular Biology and Evolution. — 2001-04-01. — Vol. 18, iss. 4. — P. 593–605. — ISSN 0737-4038 1537-1719, 0737-4038. — doi:10.1093/oxfordjournals.molbev.a003840. Архивировано 3 сентября 2022 года.
- ↑ Abdullah Ozer, Richard K Bruick. Non-heme dioxygenases: cellular sensors and regulators jelly rolled into one? (англ.) // Nature Chemical Biology. — 2007-03. — Vol. 3, iss. 3. — P. 144–153. — ISSN 1552-4469 1552-4450, 1552-4469. — doi:10.1038/nchembio863. Архивировано 3 сентября 2022 года.
- ↑ 1 2 WeiShen Aik, Michael A McDonough, Armin Thalhammer, Rasheduzzaman Chowdhury, Christopher J Schofield. Role of the jelly-roll fold in substrate binding by 2-oxoglutarate oxygenases (англ.) // Current Opinion in Structural Biology. — 2012-12. — Vol. 22, iss. 6. — P. 691–700. — doi:10.1016/j.sbi.2012.10.001. Архивировано 18 августа 2022 года.
- ↑ Zhongzhou Chen, Jianye Zang, Johnathan Whetstine, Xia Hong, Foteini Davrazou. Structural Insights into Histone Demethylation by JMJD2 Family Members (англ.) // Cell. — 2006-05. — Vol. 125, iss. 4. — P. 691–702. — doi:10.1016/j.cell.2006.04.024. Архивировано 16 июня 2022 года.
- ↑ Robert J. Klose, Yi Zhang. Regulation of histone methylation by demethylimination and demethylation (англ.) // Nature Reviews Molecular Cell Biology. — 2007-04. — Vol. 8, iss. 4. — P. 307–318. — ISSN 1471-0080 1471-0072, 1471-0080. — doi:10.1038/nrm2143. Архивировано 3 сентября 2022 года.
Ссылки
- Antiparallel β Domains, a section from Anatomy and Taxonomy of Protein Structure by Jane S. Richardson
- The Jelly Roll of Life by Jacqueline Humphries at Small Things Considered, a blog sponsored by the American Society for Microbiology
Обычно почти сразу, изредка в течении часа.

